Proteinisolation
Auswahl geeigneter Kits zur Membranproteinisolation
 Plasmamembran (PM)-Proteine spielen eine entscheidende Rolle in einer Vielzahl physiologischer und pathologischer Prozesse. Signaltransduktion, molekularer Transport und Zell-Zell-Interaktionen werden alle durch PM-Proteine vermittelt. PM-Proteine umfassen eine Vielzahl wichtiger Proteine wie Neurotransmitter-Rezeptoren, G-Proteine, Carrier, spannungsabhängige Ionenkanäle, CD-Antigene und viele Zielproteine für Medikamente. Der Nachweis, die Charakterisierung und der intrazelluläre Transport von PM-Proteinen sind daher für das Verständnis biologischer Systeme unerlässlich.
Plasmamembran (PM)-Proteine spielen eine entscheidende Rolle in einer Vielzahl physiologischer und pathologischer Prozesse. Signaltransduktion, molekularer Transport und Zell-Zell-Interaktionen werden alle durch PM-Proteine vermittelt. PM-Proteine umfassen eine Vielzahl wichtiger Proteine wie Neurotransmitter-Rezeptoren, G-Proteine, Carrier, spannungsabhängige Ionenkanäle, CD-Antigene und viele Zielproteine für Medikamente. Der Nachweis, die Charakterisierung und der intrazelluläre Transport von PM-Proteinen sind daher für das Verständnis biologischer Systeme unerlässlich.
Die Isolierung/Reinigung ist normalerweise der erste Schritt zur Charakterisierung und Profilierung von PM-Proteinen. Es hat sich jedoch gezeigt, dass dies aufgrund ihrer geringen Abundanz und der Art der Interkonnektivität der intrazellulären Membransysteme eine besondere Herausforderung darstellt. PM-Proteine werden traditionell durch Saccharosedichte-Ultrazentrifugation isoliert [1,2]. Das Protokoll ist mühsam und zeitaufwendig und dauert Stunden bis sogar Tage. In den letzten Jahren wurden in immer mehr Publikationen kommerzielle Kits für die Isolierung und Charakterisierung von PM-Proteinen zitiert, da sie einfach zu bedienen und schnell sind. Verschiedene Kits beschäftigen jedoch unterschiedliche Wirkmechanismen. Die Wirksamkeit des Kits variiert erheblich in Abhängigkeit von den spezifischen Downstream-Anwendungen. Aufgrund der Verfügbarkeit einer Vielzahl von PM-Protein-Isolierungs-/Reinigungskits auf dem Markt ist es manchmal schwierig oder sogar verwirrend, ein geeignetes Kit für ein bestimmtes Forschungsprojekt auszuwählen. In diesem Zusammenhang versuchen wir, die Vor- und Nachteile einiger häufig verwendeter Membranprotein-Isolierungskits zusammenzufassen und einen allgemeinen Leitfaden für die Auswahl kommerzieller Membranprotein-Isolierungskits bereitzustellen.
Generell lassen sich alle kommerziell erhältlichen Kits zur Isolierung von Membranproteinen nach ihren Wirkmechanismen bzw. Isolierungsprinzipien in vier grundlegende Kategorien einteilen.
- Phasen-Extraktion. Mem-PER™ Plus Membrane Protein Extraction Kit (Thermo Fisher), Mem-PER® Eukaryotic Membrane Protein Extraction Reagent Kit (Thermo Fisher) und ProteoExtract™ Native Membrane Protein Extraction Kit (Millipore-Sigma) sind typische Kits, die zu dieser Klasse gehören. Zellen/Gewebe werden zunächst mit Lysispuffer lysiert, lösliche Proteine und unlösliche Fraktionen werden durch Zentrifugation getrennt. Die Membranproteine werden anschließend mit einem Extraktionspuffer, der auf der Hydrophobizität der Membranproteine basiert, aus der unlöslichen Fraktion extrahiert. Diese Kits sind relativ einfach und schnell (ca. 1h). Das Protokoll erzeugt zwei unterschiedliche Fraktionen: die zytosolische Fraktion und die Membranproteinfraktion, jedoch geht aus dem Protokoll nicht hervor, ob ein Detergens für die Zelllyse verwendet wird. Die extrahierten Membranproteine stammen aus allen Membransystemen wie Mitochondrien, ER, Golgi und Zellkernen. Was mit diesen Kits extrahiert wird, sind eigentlich totale Membranproteine. Es kann davon ausgegangen werden, dass die Membranproteinextraktion für bestimmte Proben vollständig oder unvollständig sein kann, insbesondere für solche Proteine, die die Biomembran mehrfach durchqueren. Es ist nicht klar, ob membranassoziierte Proteine in den extrahierten Membranproteinen gebunden und intakt bleiben.
- Markierung der Zelloberfläche. Das Pierce-Kit zur Isolierung von Zelloberflächenproteinen (Thermo Fisher) und das Qproteomic Plasma-Membran-Kit (Qiagen) gehören zu dieser Klasse. Ein typisches Experiment mit dem Pierce Zelloberflächenprotein-Isolierungskit beinhaltet die Markierung von Zelloberflächenproteinen mit Sulfo-NHS-Biotin. Nach der Markierung werden die Zellen lysiert und das Zelllysat wird auf eine Avidin-konjugierte Festphase aufgetragen. Biotinylierte Plasmamembranproteine werden mit einem denaturierenden Elutionspuffer eluiert. Theoretisch sollte dieser Ansatz hoch gereinigte Plasmamembranproteine produzieren. In der Realität leidet diese Methode jedoch unter vielen ererbten Nachteilen. Zum Beispiel wird nicht jedes Protein auf der Zelloberfläche markiert werden. Das Proteinprofil einer gegebenen Zellkultur kann sich mit der Kulturzeit und den Kulturbedingungen ändern. Sterische Hinderungsgründe und fehlende primäre Amine können die Proteinmarkierung beeinträchtigen, was zu inkonsistenten Ergebnissen führt. Die Verwendung von denaturierter Elutionslösung schränkt die Anwendung von isolierten Proteinen ebenfalls ein.
- Phasenaufteilung. Ein typisches Beispiel für diese Klasse ist das Plasma Membrane Protein Extraction Kit von Biovision und Abcam. Der Wirkmechanismus ähnelt einer früher veröffentlichten wässrigen Zwei-Phasen-Partitionierungsmethode [3]. Das Protokoll nutzt den Vorteil der differentiellen Partitionierung der Plasmamembran in der oberen Phase und anderer Membranen (wie ER, Golgi und Mitochondrien) in der unteren Phase zur Anreicherung und Isolierung von Plasmamembranproteinen. Kultivierte Zellen/Gewebeproben werden zunächst mit einem Dounce-Homogenisierer homogenisiert. Die lysierten Proben werden einer mehrfachen Extraktion und Zentrifugation unterzogen, wodurch Gesamtmembranprotein, zytosolische und Plasmamembranproteinfraktionen erhalten werden. Die Vorteile der Methode bestehen darin, dass sie Detergenz-freie Plasmamembranproteine isolieren kann und das Protokoll relativ probenschonend und schnell ist (ca. 1 -1,5 h). Allerdings werden große Zellzahlen benötigt (50-100 Millionen/Probe) und die Ausbeute ist relativ gering (1-100ug/Probe). Der Hersteller behauptet, dass die Reinheit des Plasmamembranproteins über 90 % liegt, aber es werden keine unterstützenden Daten gezeigt.
- Zentrifugen-Säulen-basierte subzelluläre Fraktionierung. Dies ist eine Technologie der nächsten Generation zur Isolierung von Plasmamembranproteinen von Invent Biotechnologies, die eine einfache und schnelle Methode zur subzellulären Fraktionierung ohne Verwendung eines Dounce-Homogenisators bietet. Zellen/Gewebe werden durch eine spezielle Filterkartusche geleitet. Die Plasmamembranen der Zellen werden während des Prozesses aufgerissen und intakte Kerne, Organellen, Plasmamembran und zytosolische Proteine werden in eine Suspension freigesetzt, die weiter in fünf Fraktionen getrennt wird: Gesamtmembran-, Plasmamembran-, zytosolische, organellare und Kernfraktion. Durch die Verwendung der Filterkartusche und eines einzigartigen Puffersystems kann eine hohe Ausbeute an nativem Plasmamembranprotein in weniger als einer Stunde mit minimalen Kreuzkontaminationen gewonnen werden. Das isolierte native Plasmamembranprotein kann für beliebige Downstream-Experimente verwendet werden. Im Gegensatz zu allen anderen oben beschriebenen Kits kann das Spin-Säulen-basierte Plasmamembran-Isolierungskit auch für die Isolierung von Pflanzen-PM-Protein verwendet werden. Dieses Kit erfreut sich immer größerer Beliebtheit, wie die Daten ausgewählter Publikationen unten zeigen.
Die Auswahl eines bestimmten Membranprotein-Isolierungskits hängt hauptsächlich von den spezifischen Downstream-Anwendungen ab. Einige PM-Isolierungskits haben viel breitere Anwendungen als andere. Detergensfreie native PM-Proteine können z. B. für fast alle möglichen Downstream-Anwendungen verwendet werden, während die mit dem Pierce Zelloberflächenprotein-Isolierungskit isolierten Proteine nur für Western Blotting empfohlen werden. Die folgenden Beispiele veranschaulichen, was von einem guten subzellulären Fraktionierungskit erwartet wird. Viele PM-Proteine sind in relativ geringer Konzentration vorhanden. Manchmal ist es schwierig, Membranproteine ohne Isolierung und Anreicherung nachzuweisen und zu quantifizieren. Die folgenden Daten [4] zeigen deutlich den Effekt der PM-Protein-Anreicherung auf die Nachweisbarkeit von SGLT1 aus menschlichen Herzproben. Die Signale von SGLT und dem Plasmamembran-Markerprotein Na+/K+ ATPase sind in der Plasmamembranfraktion im Vergleich zur Gesamtmembranfraktion deutlich erhöht.

Kutluay et al. [5] untersuchten die Veränderungen der Gag-RNA-Bindung an virale RNA während der HIV-1-Virion-Assemblierung. Eines der wichtigsten Experimente ist der Nachweis unterschiedlicher Formen von Gag-RNA-Addukten, die an verschiedenen subzellulären Orten vorliegen. 4SU-gefütterte 293T-Zellen wurden mit dem proviralen HIV-1-Plasmid transfiziert und mit einem Spin-Säulen-basierten Plasma-Membran-Protein-Isolationskit fraktioniert. Die zytosolischen und Plasmamembran-Fraktionen wurden einem Western Blotting und einer Immunpräzipitation unterzogen. Die Ergebnisse zeigen, dass das zytosolische Gag-Protein hauptsächlich monomer ist, während das Gag-Protein auf der Plasmamembran multimerisiert ist (B). Diese Schlussfolgerung hängt stark von der klaren Trennung der zytosolischen und der Plasmamembranfraktionen ab (A). Wenn eine signifikante Kreuzkontamination vorhanden ist, wäre die Schlussfolgerung fragwürdig.

Untersuchungen der intrazellulären Zielproteinverteilung und des Zielprotein-Traffics unter verschiedenen Versuchsbedingungen und unterschiedlicher Behandlung von Versuchsgruppen sind für die biomedizinische Forschung wichtig. Das Zielprotein kann durch subzelluläre Fraktionierung gefolgt von einer spezifischen Nachweismethode verfolgt werden. Leung et al. [6] untersuchten den Effekt von PRL-3 auf den ULBP2-Protein-Trafficking durch Zellfraktionierung. Die humane Kolonkarzinom-Zelllinie HCT 116 wurde mit 40 um RPL-3 behandelt und einer subzellulären Fraktionierung mit einem Spin-Säulen-basierten Plasmamembran-Isolationskit unterzogen. Die Ergebnisse zeigten eine klare Trennung von PM-, Organellen- und zytosolischen Fraktionen. Die Behandlung der Zelle mit der Chemikalie führt zu einer Translokation der Plasmamembran in die Organellenfraktion. Auch diese Schlussfolgerung basiert auf der klaren Trennung der unterschiedlichen subzellulären Fraktionen.

Wie von Jose et al. [7] gezeigt, kann das Spin-column PM-Isolationskit (IBT-SM-005, Invent Biotechnologies) kultivierte Endothelzellen in verschiedene subzelluläre Fraktionen fraktionieren und, was am wichtigsten ist, das isolierte PM-Protein zeigt minimale/keine Kreuzkontamination mit Organellen wie Mitochondrien, ER und Golgi.

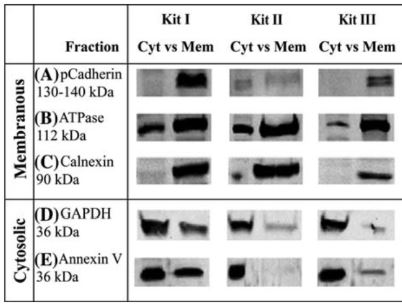
Der Grad der Kreuzkontamination ist offensichtlich einer der wichtigsten Faktoren für die Auswahl eines Kits zur Membranproteinisolierung. Die Leistung kommerzieller Kits unterscheidet sich bei diesem Kriterium erheblich. Bunger et al. [8] verglichen fünf kommerzielle Kits zur Isolierung von Membranproteinen auf Kreuzkontaminationen zwischen zytosolischen (Cyt) und Plasmamembran (Mem) Fraktionen und fanden heraus, dass alle getesteten Kits eine offensichtliche Kreuzkontamination für zwei häufig verwendete Marker (ATPase und GAPDH) aufweisen. Die Leistung der Kits variiert erheblich, wenn Cadherin als PM-Marker verwendet wird. Es wird dringend empfohlen, bei der Auswahl eines Kits zur Isolierung von Membranproteinen auf Publikationen mit hohem Impact-Faktor zurückzugreifen.

Zusammenfassend lässt sich sagen, dass Kits zur Isolierung von Membranproteinen wertvolle Werkzeuge für Forscher sind. Im Vergleich zu traditionellen Methoden kann ein guter kommerzieller Kit Zeit sparen. die Effizienz erhöhen und den Entdeckungsprozess beschleunigen. Die Auswahl eines bestimmten Kits sollte hauptsächlich auf der Leistung und den spezifischen Downstream-Experimenten beruhen. Die erforderliche Probengröße ist manchmal ein wichtiger Faktor, der berücksichtigt werden muss, insbesondere wenn die verfügbare Zellzahl ein limitierender Faktor ist. Unter idealen Bedingungen sollte ein guter kommerzieller Kit dem Forscher die erwarteten Ergebnisse ohne nennenswerte Fehlersuche und Optimierung liefern. Ein weiteres zu berücksichtigendes Kriterium ist die Frage, ob das Kit dem Forscher einen Wettbewerbsvorteil in Bezug auf Benutzerfreundlichkeit, Geschwindigkeit, Leistung und Kosten verschaffen kann.
Referenzen:
- Neville, D. M, (1960) The isolation of a cell membrane fraction from rat liver. J. Biophy. Biochem. Cyto. 8: 413-421.
- Touster, O. et al. (1970) Isolation of liver plasma membranes. J. Cell Bio. 47:604-618.
- Yoshida, S. et al. (1983) Partitioning of membrane particles in aqueous two-polymer phase system and its practical use for purification of plasma membranes from plants.Plant physiol. 72:105-114.
- Kashiwagi Y. et al. (2015) Expression of SGLT1 in Human Hearts and Impairment of Cardiac Glucose Uptake by Phlorizin during Ischemia- Reperfusion Injury in Mice. PLoS ONE 10(6):e0130605.
- Kutluay, S., et al. (2014). Global Changes in the RNA Binding Specificity of HIV-1 Gag Regulate Virion Genesis, Cell (2014), http://dx.doi.org/10.1016/j.cell.2014.09.057
- Leung W-H. et al. (2015). PRL-3 Mediates the protein Maturation of ULBP2 by regulating the tyrosine phosphorylation of HSP60.The Journal of Immunology.doi:10.4049/jimmunol.1400817.
- Vazquez-Medina J.P. et al. (2016). The phospholipase A2 activity of peroxiredoxin 6 modulates NADPH oxidase 2 activation vialysophosphatidic acid receptor signaling in the pulmonary endothelium and alveolar macrophages. The FASEB Journal. doi: 10.1096/fj.201500146R
- Bunger, S. et al. (2009) Comparison of five commercial extraction kits for subsequent membrane protein profiling. Cytotechnology. 61:153-159.